Finde den Unterschied
 Vielleicht haben sich einige gefragt „Und nun? Wie geht es jetzt weiter damit? Wozu der ganze Aufwand?“. Um ehrlich zu sein, manchmal habe ich mich das auch schon gefragt… aber dann kommen doch wieder spannende Momente, die anspornen. So wie vor 3 Wochen, als sich die „Coleanthus-Spezialisten“ aus Polen, Tschechien und Deutschland getroffen haben, um im Biosphärenreservat Oberlausitzer Heide- und Teichlandschaft zu beraten, wie die dort vorkommenden Populationen geschützt werden können. Und dabei wurde sehr deutlich, wie wichtig es ist zu wissen, wo die Art ursprünglich herstammt und wie sie sich verbreitet. Denn darüber ist bisher nicht viel bekannt.
Vielleicht haben sich einige gefragt „Und nun? Wie geht es jetzt weiter damit? Wozu der ganze Aufwand?“. Um ehrlich zu sein, manchmal habe ich mich das auch schon gefragt… aber dann kommen doch wieder spannende Momente, die anspornen. So wie vor 3 Wochen, als sich die „Coleanthus-Spezialisten“ aus Polen, Tschechien und Deutschland getroffen haben, um im Biosphärenreservat Oberlausitzer Heide- und Teichlandschaft zu beraten, wie die dort vorkommenden Populationen geschützt werden können. Und dabei wurde sehr deutlich, wie wichtig es ist zu wissen, wo die Art ursprünglich herstammt und wie sie sich verbreitet. Denn darüber ist bisher nicht viel bekannt.
Also habe ich mich mit frischem Tatendrang an die weitere Auswertung gemacht. Wie funktioniert das nun eigentlich? Es geht um eine spezielle Art des ‚genetischen Fingerabdruckes‘, um damit die Ähnlichkeit der Populationen untereinander zu bestimmen. Die Theorie dahinter besagt, dass jedes Individuum Mutationen in sich trägt, die keine Auswirkungen auf die Lebensfähigkeit haben, und dennoch weitervererbt werden – sogenannte Polymorphismen. Man geht davon aus, je länger Populationen getrennt sind bzw. je weiter sie voneinander entfernt liegen, desto stärker unterscheiden sie sich durch solche Polymorphismen. Das kann man durch die Analyse des Erbgutes – der DNA – herausfinden. Ich verwende die sogenannte AFLP-Methode (AFLP = Amplified Fragment Length Polymorphism). Dazu wird die aus den Pflanzen extrahierte DNA zunächst gezielt durch Enzyme in Stücke geschnitten. Da die Enzyme immer nur an bestimmten Nukleotid-Sequenzen schneiden können, kann sich bei vorhandenen Mutationen die Anzahl und Länge der DNA-Bruchstücke zwischen den Pflanzenindividuen unterscheiden. Die Stücke werden dann mit Hilfe der Polymerase Chain Reaction (PCR) vervielfältigt und gleichzeitig mit Farbmarkern versehen. Das Gemisch der DNA-Bruchstücke wird mittels der Gelelektrophorese der Größe bzw. Länge nach aufgetrennt. Dadurch ergibt sich ein typisches Bandenmuster. Die Anzahl der Bruchstücke je Größe kann über die Fluoreszenzintensität des Farbmarkers bestimmt werden. Dieser Schritt der Gelelektrophorese und Quantifizierung erfolgt heutzutage maschinell, also mit dem „Sequencer“. Als Ergebnis bekommt man dann je Probe vier Chromatogramme mit den verschiedenen Farbmarkern, auf denen die Bruchstücke als „Peaks“ angezeigt werden. Eine andere Bruchstücklänge bedeutet eine andere Position im Chromatogramm, und durch diese Position unterscheiden sich nun auch die Polymorphismen.
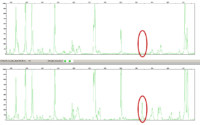 Und was kommt nun? Eine Tätigkeit in der Art „Finde den Unterschied!“. Zum Teil können da Programm-Tools beim Finden der Polymorphismen helfen, aber den ersten Durchgang muss man manuell machen. Bei 478 Proben mal 4 Farbmarkern mal durchschnittlich 60 Peaks pro Probe eine Herausforderung. An dieser Stelle bin ich jetzt gerade. Auf jeden Fall kann ich schon sagen – es gibt Unterschiede! Ob nun die Freiberger Populationen mehr denen in der Lausitz oder den russischen Pflanzen ähneln, das ist noch nicht mit Sicherheit zu sagen, aber es gibt Hoffnung auf Ergebnisse. Wenn alles gut läuft, kann man den Naturschützern aus dem Biosphärenreservat dann vielleicht sagen, woher die dortigen Coleanthus– Pflanzen eingewandert sind, und wie man den genetischen Austausch fördern kann. Ich halte euch auf dem Laufenden.
Und was kommt nun? Eine Tätigkeit in der Art „Finde den Unterschied!“. Zum Teil können da Programm-Tools beim Finden der Polymorphismen helfen, aber den ersten Durchgang muss man manuell machen. Bei 478 Proben mal 4 Farbmarkern mal durchschnittlich 60 Peaks pro Probe eine Herausforderung. An dieser Stelle bin ich jetzt gerade. Auf jeden Fall kann ich schon sagen – es gibt Unterschiede! Ob nun die Freiberger Populationen mehr denen in der Lausitz oder den russischen Pflanzen ähneln, das ist noch nicht mit Sicherheit zu sagen, aber es gibt Hoffnung auf Ergebnisse. Wenn alles gut läuft, kann man den Naturschützern aus dem Biosphärenreservat dann vielleicht sagen, woher die dortigen Coleanthus– Pflanzen eingewandert sind, und wie man den genetischen Austausch fördern kann. Ich halte euch auf dem Laufenden.
Hier geht es zum ersten Teil meines Berichts: Alles Gras oder was?


Hinterlasse einen Kommentar
An der Diskussion beteiligen?Hinterlasse uns deinen Kommentar!